生物体遗传信息DNA缠绕组蛋白八聚体1.7圈形成了染色体的基本组成单位——核小体。组蛋白H4的 N端尾巴与临近的核小体相互作用,促进染色体高级结构的形成以及异染色质沉默。核小体组装和异染色质形成阻碍了DNA的复制、转录以及损伤修复等重要生物学过程。生物体进化出了一系列的机制来克服核小体的阻碍。其中,组蛋白乙酰化修饰中和赖氨酸侧链的正电荷,并招募其他染色质因子,进而调控染色质折叠、基因转录以及DNA损伤修复等过程。
2022年10月5日,清华大学生命科学学院/结构生物学高精尖创新中心/清华-北大生命科学联合中心陈柱成教授与李雪明副教授合作在《自然》杂志在线发表题为“酿酒酵母NuA4乙酰转移酶复合物结合核小体的结构”( Structure of the NuA4 acetyltransferase complex bound to the nucleosome)的研究论文,揭示了乙酰转移酶NuA4结合核小体以及组蛋白H4空间识别的机理,阐明了NuA4作为转录共激活因子发挥功能的结构基础。
NuA4和SAGA是酿酒酵母两个重要的乙酰转移酶复合物,分别选择性地乙酰化组蛋白H4和H3,调控基因转录。NuA4和SAGA在不同物种中高度保守,前者是酵母生存唯一必需的乙酰转移酶。作为转录共激活因子,二者通过共同亚基Tra1,结合转录因子,被招募至启动子区(图1a)。因为有基础性作用,NuA4自发现至今20多年被不断研究,但其结构研究历史相当曲折,目前尚不清楚精细的分子组装模式以及识别底物核小体的机理。
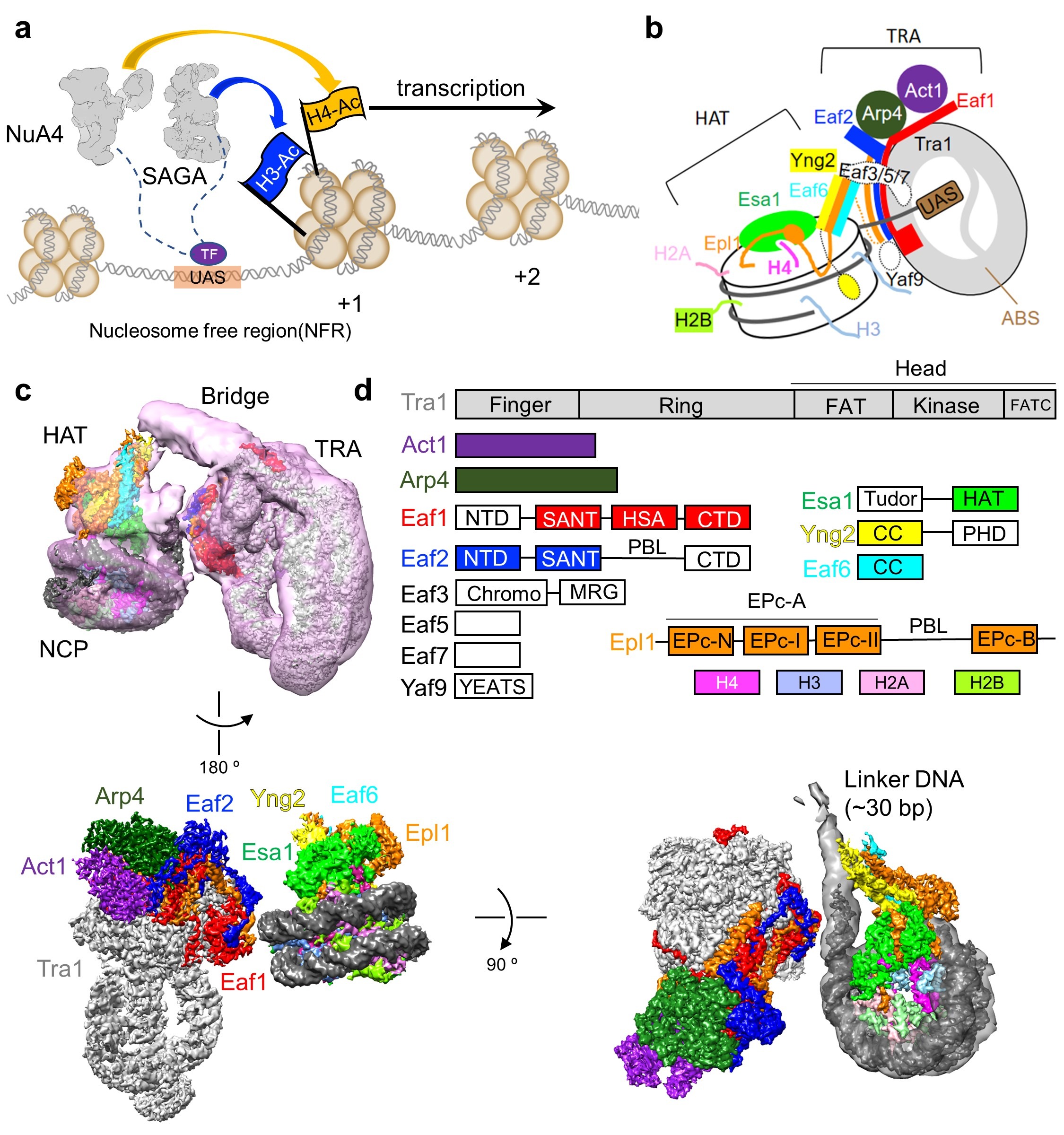
图1 NuA4的工作模式图及结合核小体的结构
(a)NuA4与SAGA协同作用促进基因转录的工作模式图;(b)NuA4结合核小体的示意图;(c)NuA4-NCP复合物的电镜密度图。(d)NuA4各亚基结构域组成示意图,颜色与电镜密度图中一致。
陈柱成课题组早期报道了NuA4活性中心Piccolo亚复合物的晶体结构及其结合核小体的低分辨率冷冻电镜结构。在此基础上,研究团队继续向着NuA4全酶的结构解析进击。然而,研究人员发现NuA4复合物的结构可塑性非常大,其活性中心结合核小体的部分无法得到稳定构象。这造成高质量结构解析的关键技术难点。为了限制复合物结构的柔性,研究团队在样品中加入了转录因子Gal4-VP16,并在核小体接头 DNA引入相应的识别DNA序列,作为上游激活信号(UAS)。而且,研究团队对组蛋白H4K16位点进行CMC(carboxymethyl coenzyme A)化学修饰以及H3K36位点进行三甲基化修饰。CMC结合NuA4的催化中心Esa1,稳定Esa1与核小体的作用;H3K36则结合Eaf3亚基。在突破各种技术难题后,最终解析了NuA4结合核小体的冷冻电镜结构(8.8 Å),局部分辨率为2.7-3.4 Å。
由13个亚基组成的NuA4复合物分为两个大的模块 (图1c):乙酰化模块(histone acetyltransferase, HAT)以及转录因子结合模块(transcription activator-binding, TRA)。HAT模块即为piccolo亚复合物,由Esa1、Epl1的N端区域、Eaf6以及Yng2组成;TRA模块则由Tra1、Eaf1、Eaf2、Act1、Arp4以及Epl1的C端区域组成。Epl1的一段无序区把HAT模块与TRA模块连接在一起。另外,还存在一个“桥型”的结构柔性区。研究人员根据交联质谱推断这部分区域为Eaf3/5/7亚基。从这里可以看出NuA4复合物构造的高度可塑性。
该结构显示,携带UAS的linker DNA延伸向Tra1亚基表面(图2a)。研究人员发现该表面在不同物种中高度保守(图2b),且众多转录因子与Tra1的结合位点均位于该表面内。因此,该保守面被命名为转录因子结合面(transcription factor binding surface, ABS)。ABS的发现提供了NuA4被转录因子招募至启动子区域的结构基础。
在ABS外周,研究人员发现一个多碱性氨基酸界面(poly-basic surface, PBS)与核小体的linker DNA相互作用(图2c)。研究人员通过生化以及遗传学实验验证了PBS的对乙酰转移活性,以及调控酵母碳源代谢的重要性(图2d)。
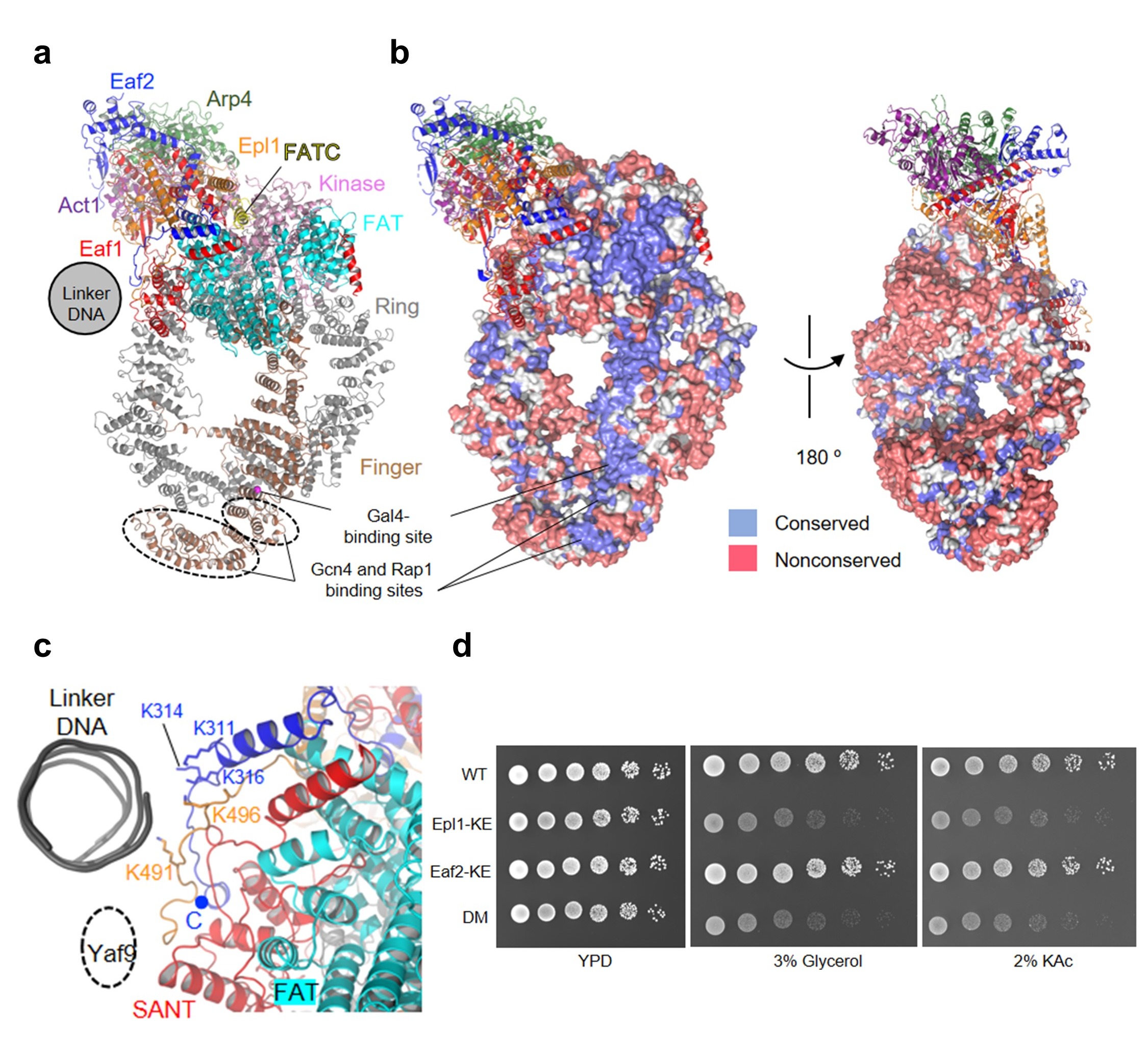
![]() 图2 NuA4的 TRA模块结构和核小体识别机理
图2 NuA4的 TRA模块结构和核小体识别机理
(a)转录因子结合表面(ABS); (b) ABS的保守性分析(c)多碱性氨基酸表面(PBS)的结构; (d) 野生型和PBS突变体的生长表型分析。
催化中心HAT模块通过两个关键元件结合在核小体盘状结构表面。其中,Epl1的“arginine anchor”识别核小体的酸性区,“double function loop (DFL)”识别核小体的超螺旋1.5位置处DNA(图3a, b, c)。该核小体识别模式使得Esa1的活性口袋恰好处在H4 的N端尾巴上方。这个结构在全酶水平支持了H4尾巴空间位置识别机制(图3b)。该机制区别于常见的基于氨基酸序列识别的组蛋白修饰酶工作机制。
综上所述,该研究揭示了NuA4通过多个结构元件,协调识别核小体的机理(图1b)。ABS通过转录因子,PBS直接结合接头DNA,招募核小体至TRA模块边缘;此构象使得HAT模块通过DFL和arginine anchor识别核小体的表面,从而发挥选择性乙酰化H4的功能。TRA模块与HAT模块之间的可塑性,使得NuA4能够适应复杂的染色体环境,被不同转录因子招募。除了Eaf5亚基之外,其他亚基在人源TIP60复合物中均存在同源蛋白。因此,该研究对于理解TIP60复合物的组装及工作机制提供了很好的模型。
值得一提的是,该工作TRA模块的结构被另外三个单独的NuA4复合物的结构研究所验证(预印版,见延伸阅读)。
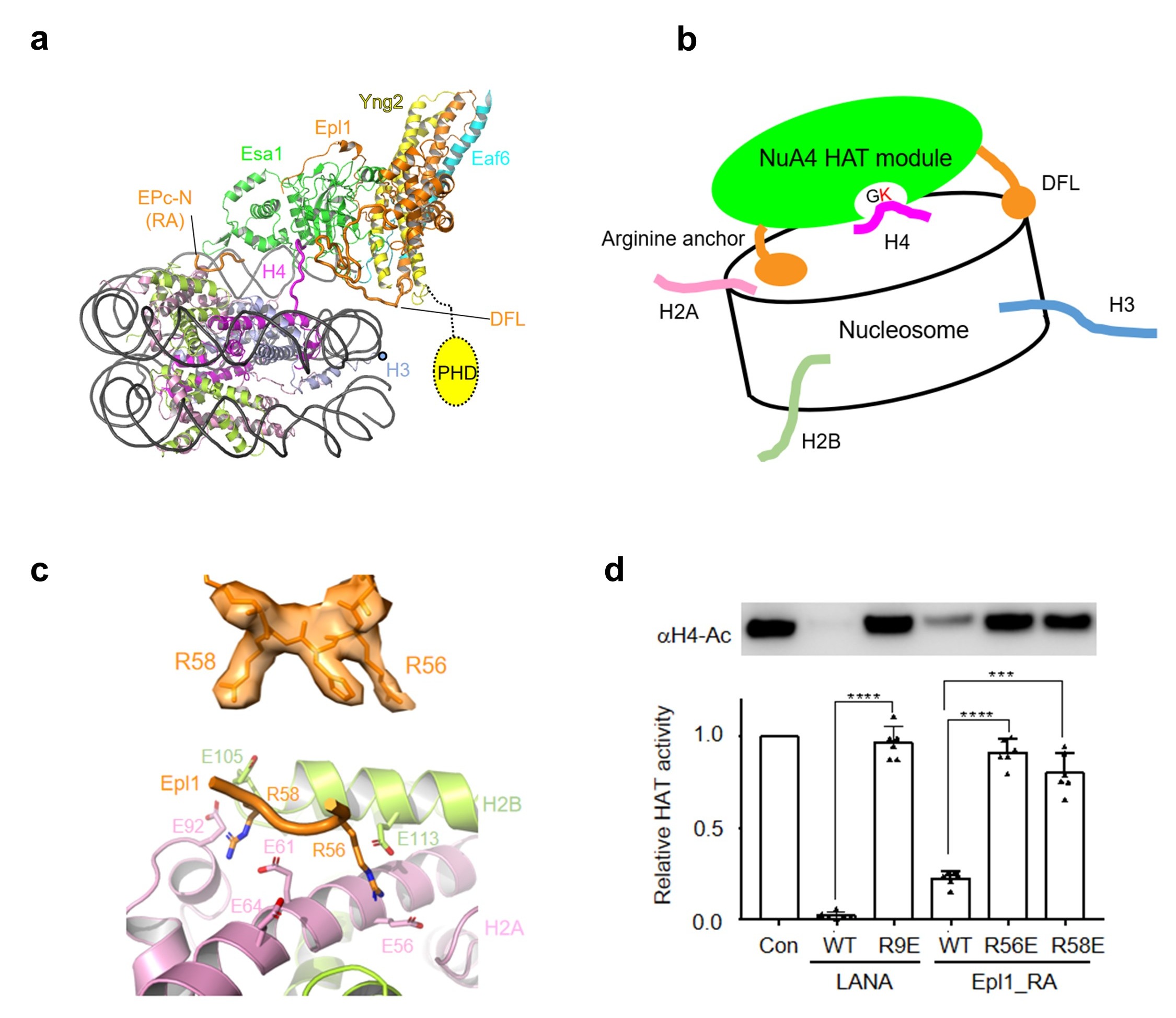
图3 NuA4 HAT模块识别核小体的机理
(a)HAT模块结合核小体的结构模型;(b)NuA4通过空间位置识别H4的模式图;(c)Arginine anchor局部电镜密度图及其与酸性区相互作用结构细节;(d)多肽竞争性实验显示Arginine anchor对NuA4乙酰化活性的重要性。
清华大学生命科学学院陈柱成教授、李雪明副教授为本文共同通讯作者,清华大学生命科学学院2014级博士生瞿珂珂(已毕业)和2017级博士生陈康净(已毕业)、2017级博士生王皓为该论文共同第一作者。本工作获得国家自然科学基金、科技部重大科学研究计划专项、北京市结构生物学高精尖创新中心、清华-北大生命科学联合中心、国家蛋白质科学研究(北京)设施清华基地的大力支持,国家蛋白质科学研究(北京)设施清华基地冷冻电镜平台和计算平台为数据收集和处理提供了支持。
原文链接:https://www.nature.com/articles/s41586-022-05303-x
延伸阅读:
https://www.researchsquare.com/article/rs-1497616/v1
https://www.biorxiv.org/content/10.1101/2022.07.11.499577v1
https://www.biorxiv.org/content/10.1101/2022.06.24.497536v1
